I3D:bio
Information Infrastructure for BioImage Data

Die Mikroskopie umfasst eine Vielzahl von Methoden zur Gewinnung von räumlich und zeitlich hoch aufgelösten Forschungsdaten und kommt in allen Bereichen der Lebenswissenschaften und Medizin zum Einsatz. Verschiedene Verfahren erlauben es, mithilfe von Licht- oder Elektronenstrahlen Bilddaten zu erzeugen. Diese Daten werden als Mikroskopiebilder visualisiert und aus Ihnen können mithilfe von Bildanalyse quantitative Informationen über das Untersuchungsobjekt erhalten werden. Durch die Probenvorbereitung können gezielt bestimmte Strukturen sichtbar gemacht und gemessen werden. Beispielmethoden sind etwa die konfokale Lasermikroskopie mit Fluoreszenszfarbstoffen oder Auflicht- und Durchlichtmikroskopie im Weitfeld mithilfe von verschiedenen Färbeverfahren. Nicht nur liefern Verfahren wie Hochdurchsatzmikroskopie oder spezielle Techniken wie die Lichtblattmikroskopie sehr große Datenmengen. Die entstehenden Bilddateien sind zudem oft komplex strukturiert (4-, 5-, oder mehrdimensional) und können sehr große Einzeldateigrößen erreichen (GB bis TB). Zugleich sind die Dateiformate und Metadaten-Strukturen der verschiedenen Gerätehersteller bis heute wenig standardisiert.
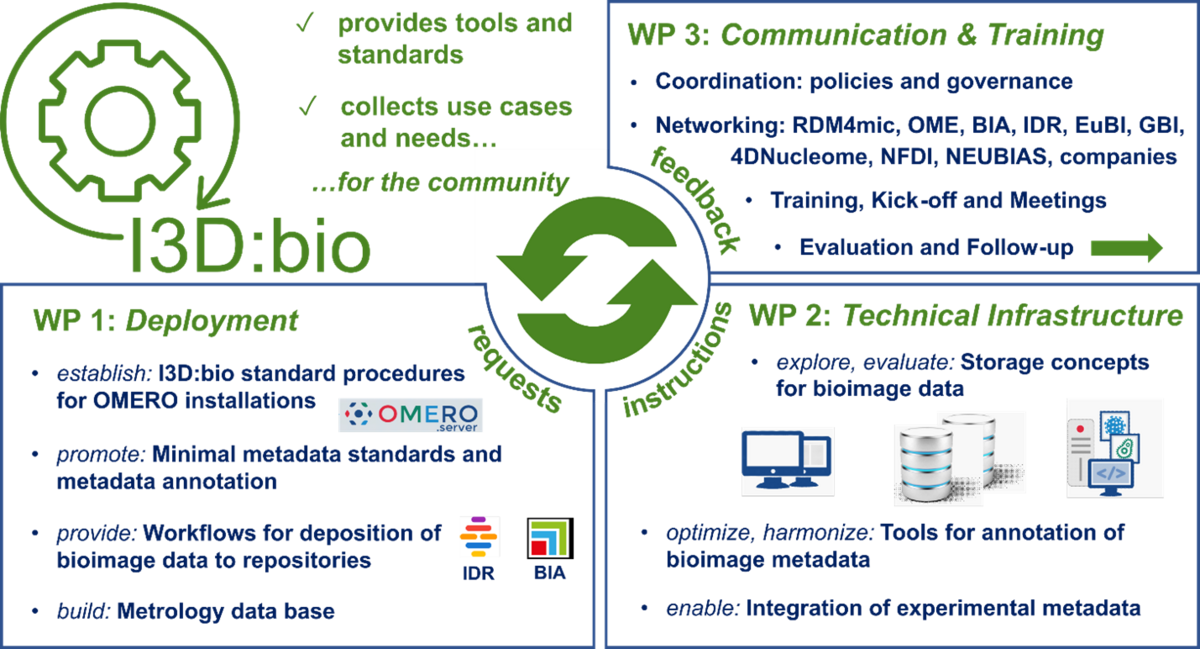
Vor diesem Hintergrund stellt das Forschungsdatenmanagement im Bereich Mikroskopie besondere Herausforderungen and Forschende. Das von der Deutschen Forschungsgemeinschaft (DFG) geförderte Projekt Information Infrastructure for BioImage Data (I3D:bio) befasst sich mit Lösungen für das Management und das Teilen von Forschungsdaten im Bereich Bioimaging. Ein Schwerpunkt des Projekts ist die unterstützte Implementation der offenen Software OMERO (OME Remote Objects), die vom Open Microscopy Environment Consortium (OME) speziell für das Bilddatenmanagement entwickelt wurde. Das Projekt I3D:bio erarbeitet zielgruppengerechte Installationshilfen und unterstützt Partnerinstitutionen an verteilten Standorten in Deutschland bei der Einführung von OMERO. Insbesondere die Lichtmikroskopie-Gerätezentren sollen auf diese Weise ihre Nutzenden besser beim Bilddatenmanagement unterstützen können. Darüber hinaus erarbeiten die Projektpartner neue Lösungen für die Interoperabilität von OMERO mit elektronischen Laborbüchern und Empfehlungen für standardisierte Metadaten-Annotation in Schlagworten und Key-Value-Paaren in OMERO. Die unmittelbaren Vorteile für Nutzende der OMERO Software werden herausgestellt und es werden Trainingsmaterialien für den Umgang mit OMERO erarbeitet. Durch ein verbessertes Management der Bilddaten von Anfang an soll zudem die Ablage publizierter Daten in Bilddatenrepositorien wie dem BioImage Archive oder der Image Data Resource erleichtert werden.
In einem weiteren Teilprojekt soll eine bessere Vergleichbarkeit der Qualitätskontrollen an Mikroskopen vor der Bildakquisition ermöglicht werden. Hierzu wird ein Repositorium auf der Basis von OMERO geschaffen. Eine bessere Vergleichbarkeit der Protokolle beim Management der Geräte erhöht die Qualität der entstehenden Bilddaten und schafft eine Grundlage für Vergleichbarkeit öffentlich geteilter Forschungsdaten aus der Mikroskopie. Das Ziel des Projektes insgesamt ist es, die Implementation der FAIR-Prinzipien Auffindbarkeit, Zugriff, Interoperabilität und Nachnutzbarkeit für biologische Bilddaten im praktischen Forschungsalltag voranzubringen.
Services
- Help Desk für die Implementation von OMERO an Universitäten und Forschungseinrichtungen
- Metrologie-Datenbank (im Aufbau)
- Knowledge-Hub für Forschungsdatenmanagement mit biologischen Bilddaten
- Training für Bilddatenmanagement mit OMERO
Projektpartner*innen
- Heinrich-Heine-Universität Düsseldorf, Center for Advanced Imaging
- Deutsches Krebsforschungszentrum, Abt. Enabling Technology
- Universität Osnabrück, Integrated Bioimaging Facility Osnabrück, CellNanOs
- Albert-Ludwigs-Universität Freiburg, Life Imaging Center
- Unterstützt von German BioImaging – Gesellschaft für Mikroskopie und Bildanalyse
- I3D:bio arbeitet eng mit den NFDI-Konsortium NFDI4BIOIMAGE zusammen
Projektlaufzeit
01.01.2022 – 31.12.2024
Projektförderung
Deutsche Forschungsgemeinschaft (DFG), Projektnummer 462231789
Veröffentlichungen
- I3D:bio's OMERO Training Videos 2023, https://www.youtube.com/playlist?list=PL2k-L-zWPoR7SHjG1HhDIwLZj0MB_stlU, based on: Schmidt, C., Bortolomeazzi, M., Boissonnet, T., Fortmann-Grote, C., Dohle, J., Zentis, P., Kandpal, N., Kunis, S., Zobel, T., Weidtkamp-Peters, S., & Ferrando-May, E. (2023). I3D:bio's OMERO training material: Re-usable, adjustable, multi-purpose slides for local user training. Zenodo. https://doi.org/10.5281/zenodo.832358
- Kunis, Susanne, & Dohle, Julia. (2022). Structuring of Data and Metadata in Bioimaging: Concepts and technical Solutions in the Context of Linked Data. Zenodo. https://doi.org/10.5281/zenodo.7018750
- Schmidt C, Hanne J, Moore J et al. Research data management for bioimaging: the 2021 NFDI4BIOIMAGE community survey. F1000Research 2022, 11:638 (https://doi.org/10.12688/f1000research.121714.2)
- Schmidt, C., Bortolomeazzi, M., Boissonnet, T., Fortmann-Grote, C., Dohle, J., Zentis, P., Kandpal, N., Kunis, S., Zobel, T., Weidtkamp-Peters, S., & Ferrando-May, E. (2023). I3D:bio's OMERO training material: Re-usable, adjustable, multi-purpose slides for local user training. Zenodo. https://doi.org/10.5281/zenodo.8323588
- Schmidt, Christian et al.: Bringing FAIR Bioimage Data Management into Practice: the Information Infrastructure for BioImage Data (I3D:bio) Project – bottom-up Community Support for Microscopy Data Sharing and Preservation., in Heuveline, Vincent, Bisheh, Nina und Kling, Philipp (Hrsg.): E-Science-Tage 2023: Empower Your Research – Preserve Your Data, Heidelberg: heiBOOKS, 2023, S. 289–294. https://doi.org/10.11588/heibooks.1288.c18090
Weitere Informationen
- Webseite und Knowledge Hub
- Reguläre Treffen der Arbeitsgruppe RDM4mic (Research Data Managment for Microscopy), aus der das I3D:bio Projekt hervorgegangen ist
- German BioImaging
Kontakt
Kontakt über die Projektwebseite: https://gerbi-gmb.de/i3dbio/i3dbio-help-contact/
Email: i3dbio@gerbi-gmb.de
